Universidad e investigación
Servicios de secuenciación de ADN exclusivos para académicos e investigadores
Neoresearch
Neoprospecta es una empresa con origen en el ámbito académico, lo que refuerza nuestro compromiso con el avance de la investigación científica en nuestro ADN.
Mantenemos una política activa de apoyo a la investigación, ofreciendo análisis moleculares y servicios de bioinformática con precios accesibles, entrega ágil y sin complicaciones.
Todo esto respaldado por un equipo técnico altamente capacitado y con acceso a equipos de última generación, garantizando el rigor científico que su experimento requiere.
Nuestras plataformas:
- MiSeq – Illumina
- Next-Seq – Illumina
- Minion – Nanopore Oxford
Nosso WorkFlow
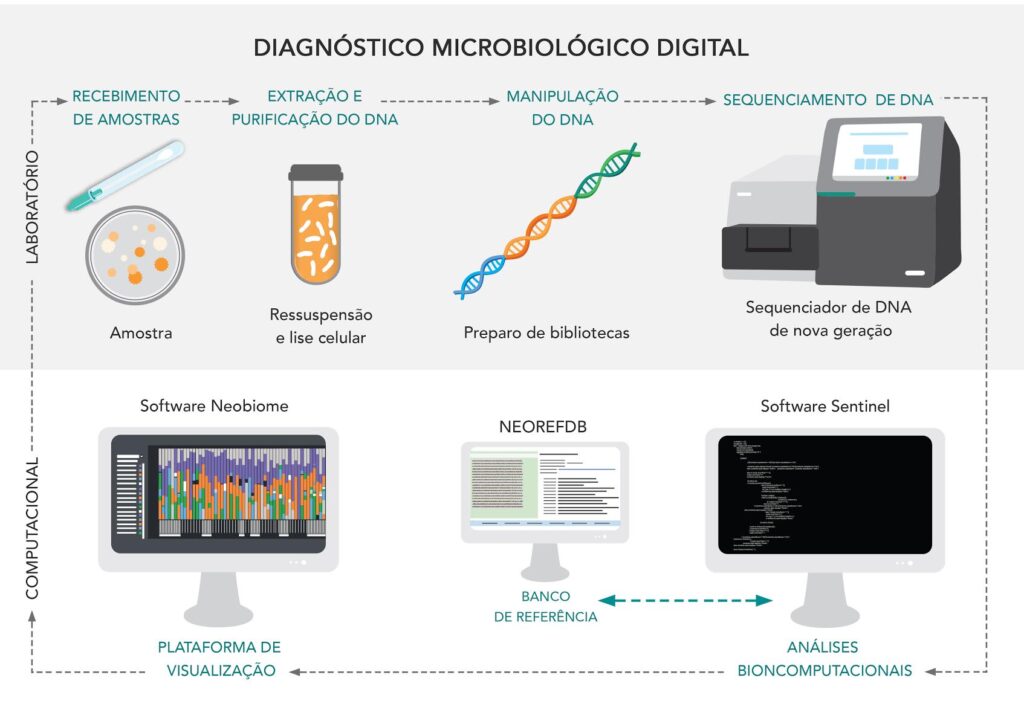
Nuestro servicio incluye: extracción de ADN, preparación de bibliotecas, secuenciación a gran escala y análisis básico de bioinformática para la clasificación taxonómica de los reads generados. Entregamos los datos brutos de la secuenciación (FastQ), la tabla de clasificación (ranktable) y el acceso a la Plataforma Neobiome.
Nuestros análisis
NGS – Secuenciación de amplicones 16S
Utiliza la región V3/V4 del rRNA 16S para identificar la microbiota bacteriana presente en muestras complejas o aisladas. Puede aplicarse en diversas áreas de investigación como estudios de diversidad en ecosistemas, análisis de microbiota animal, desarrollo de nuevos productos e investigaciones microbiológicas en alimentos, entre otros.
NGS – Secuenciación de amplicones ITS
Utilizando la técnica de metabarcoding, la secuenciación del ADN de nueva generación dirigida a la región ITS permite identificar a gran escala hongos presentes en diversos tipos de muestras, como suelos, alimentos, lodos, aguas residuales, entre otros que sean de interés del investigador.
NGS – Secuenciación de amplicones 18S
Mediante el uso de cebadores específicos para el gen 18S rRNA, este análisis de secuenciación de ADN permite el screening de una amplia diversidad de protozoarios. Esta metodología molecular puede ser empleada para la caracterización de parásitos en diversos tipos de muestras, estudios de biodiversidad, sanidad animal e investigaciones alimentarias, entre otros.
WGS – Secuenciación de genoma completo
Diseñado para satisfacer demandas específicas y complejas, también ofrecemos el servicio de secuenciación del genoma completo de aislados bacterianos y fúngicos mediante el enfoque shotgun.
Utilizando plataformas Illumina NextSeq o MiSeq, brindamos diversas opciones de cobertura y entregas bioinformáticas personalizadas para que tenga total libertad de elección, basada en el objetivo clave de su investigación.
WGS – Metagenómica
Mediante el enfoque shotgun, ofrecemos análisis de metagenómica en diferentes tipos de muestras.
A través de las plataformas NextSeq o MiSeq es posible realizar el servicio en diferentes coberturas.
Entregamos los datos brutos de secuenciación (FastQ) y podemos realizar análisis de bioinformática específicos de acuerdo con la necesidad del cliente.
Bioinformática
El equipo de bioinformática es responsable de recolectar, procesar, analizar e interpretar datos biológicos, especialmente aquellos relacionados con secuencias de ADN.
Análisis de Datos Genómicos:
Interpretan secuencias de ADN, identificando genes, variantes genéticas y características específicas de los organismos, como patógenos en alimentos o características genéticas.
Ejemplos:
Secuenciación de amplicones – identificación taxonómica
Genoma Completo – ensamblaje y anotación de genes
Serotipado
Clonalidad
Diversidad Alfa y Beta


Dúvidas frequentes:
Amplicón:
Objetivo: Identificar todos los organismos presentes en la muestra, utilizando marcadores genéticos.
Tipo de objetivo: Regiones altamente conservadas del ADN (por ejemplo, 16S) que identifican géneros o especies.
Ventajas: Mayor número de objetivos, revelando diferencias entre grupos de muestras.
Técnica: Amplificación de marcadores y secuenciación.
Tipo de resultado: Abundancia relativa de microorganismos.
Enriquecimiento: Depende del ensayo.
Genoma Completo:
Objetivo: Identificar especies/aislados a nivel de genoma completo.
Tipo de objetivo: Todo el genoma del microorganismo.
Ventajas: Enfoque robusto para la identificación y caracterización de aislados.
Técnica: Fragmentación, secuenciación y ensamblaje del genoma.
Tipo de resultado: Caracterización de especie, resistencia a antibióticos, serotipado, clonalidad.
Enriquecimiento: No es necesario.
PCR:
Objetivo: Detectar un organismo específico (por ejemplo, Salmonella).
Tipo de objetivo: Gen o secuencia específica.
Ventajas: Máxima sensibilidad en muestras complejas.
Técnica: PCR en Tiempo Real de marcadores específicos.
Tipo de resultado: Presencia/ausencia del microorganismo objetivo.
Enriquecimiento: Necesario en algunos casos (Listeria, Salmonella, etc.).
La secuenciación de ADN a gran escala realizada por Neoprospecta permite identificar la diversidad de bacterias y/u hongos en una muestra mediante el análisis de las regiones 16S rRNA y/o ITS rRNA. Esta metodología proporciona una visión integral de los microorganismos presentes, utilizando un pipeline bioinformático que optimiza la resolución de los datos.
Los resultados presentan alta consistencia y reproducibilidad, independientemente de las matrices técnicas analizadas, garantizando perfiles microbiológicos confiables.
En contrataciones puntuales (spot), los datos del perfil microbiológico —herramienta dentro de la Plataforma Neobiome— están disponibles por un período de 6 meses. Los gráficos generados por esta herramienta pueden ser ampliamente utilizados para la evaluación de resultados y generación de conocimiento, y el BIM – Banco de Información de Microorganismos puede ser contratado para el análisis detallado de las características de los microorganismos identificados.
Los datos brutos están disponibles por aproximadamente 90 días a partir del envío del enlace de descarga. Se recomienda que el cliente realice la descarga lo antes posible, ya que una vez vencido este plazo, los datos se eliminan. En caso de expiración, es posible solicitar la generación de un nuevo enlace mediante una nueva propuesta comercial, debido a los costos involucrados.
La identificación a nivel de género ocurre cuando múltiples especies presentan similitudes en las secuencias, lo que imposibilita una especificación precisa.
Sin embargo, Neoprospecta ha desarrollado otras pruebas que utilizan primers dirigidos a blancos específicos, aumentando así la resolución a nivel de especie.
La principal ventaja de utilizar primers genéricos como el 16S es que las muestras con microbiota desconocida, es decir, "a ciegas", permiten una caracterización general del conocimiento sobre la composición microbiológica de la muestra, lo cual es fundamental para la toma de decisiones.
Muestras con sospecha previa de blancos bacterianos o fúngicos deben informarse al equipo comercial para seleccionar los ensayos más adecuados según el objetivo de la evaluación.
Neoprospecta utiliza sus propias bases de datos validadas para los análisis de secuenciación, desarrolladas por el equipo de bioinformática a partir de bases públicas como GenBank y SILVA. En estas bases, las secuencias con errores o quimeras son eliminadas.
Para análisis bacterianos (gen 16S rRNA), también están disponibles las bases públicas SILVA y Greengenes, si el cliente así lo solicita. Sin embargo, dichas bases públicas no pasan por la curaduría de Neoprospecta, por lo que la empresa no garantiza la calidad de los resultados cuando estas son utilizadas.

