Universidade e pesquisa
Serviços de sequenciamento de DNA exclusivo para acadêmicos e pesquisadores
Neoresearch
A Neoprospecta é uma empresa que surgiu do meio acadêmico, por isso trazemos o comprometimento com a pesquisa brasileira em nosso DNA.
Seguindo uma política de apoio e incentivo a pesquisa, disponibilizando análises moleculares e serviços de bioinformática com valores acessíveis, de forma rápida e descomplicada.
Tudo isso contando com o apoio de um time de profissionais experientes e equipamentos de ultima geração, entregando o rigor técnico científico que seu experimento precisa.
Nossas plataformas:
- MiSeq – Illumina
- Next-Seq – Illumina
- Minion – Nanopore Oxford
Nosso WorkFlow
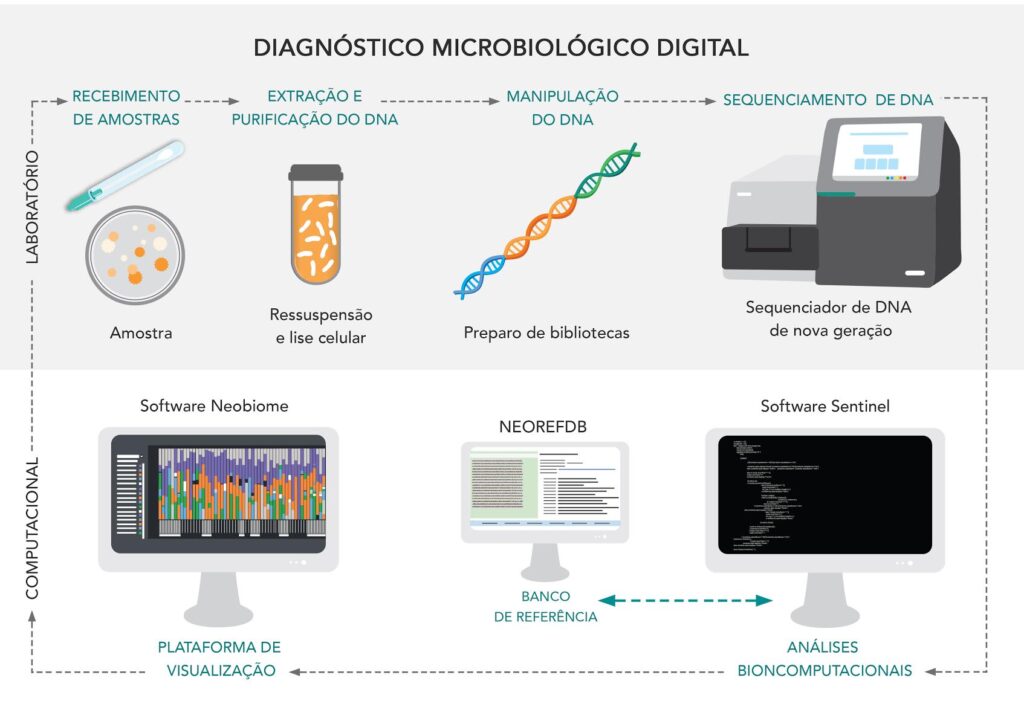
Nosso serviço engloba desde extração de DNA, preparo das bibliotecas, sequenciamento em larga escala e análise de bioinformática básica para a classificação taxonômica dos reads gerados. Entregamos os dados brutos do sequenciamento (FastQ), ranktable e o acesso a Plataforma Neobiome.
Nossas análises
NGS - Sequenciamento de amplicons 16S
Utiliza a região V3/V4 do rRNA 16S para identificar a microbiota bacteriana presente em amostras complexas ou isolados podendo ser utilizado em diversas áreas da pesquisa como estudos de diversidade em ecossistemas, investigação da microbiota animal, desenvolvimento de novos produtos, investigação microbiológicas em alimentos entre outras.
NGS - Sequenciamento de amplicons ITS
Utilizando a técnica de metabarcoding, o sequenciamento de DNA de nova geração da região ITS permite identificar em larga escala os fungos presentes em diferentes tipos de amostras como isolados, alimentos, lodo, solo, água, fezes, entre outras que sejam de interesse do pesquisador.
NGS - Sequenciamento de amplicons 18S
Utilizando primers para o gene 18S rRNA está análise de sequenciamento de DNA pode ser utilizada para screeaning de uma ampla variedade de protozoários. Essa método molecular pode ser utilizado para caracterização de parasitos em uma variedade de amostras e estudos de biodiversidade, sanidade animal, investigações em alimentos, entre outros.
WGS - Sequenciamento de genoma completo
Projetado para atender demandas específicas e complexas disponibilizamos também o serviço de sequenciamento do genoma completo de isolados bacterianos e fúngicos utilizando a abordagem shotgun.
Utilizando a plataforma Illumina NextSeq ou MiSeq. oferecemos diversas opções de cobertura e entregas de bioinformática customizadas para que você tenha total liberdade de escolha com base no objetivo chave da sua pesquisa.
WGS - Metagenômica
Utilizando a abordagem shotgun oferecemos análises de metagenômica em diferentes tipos de amostras.
Por meio das plataformas Next-seq ou MiSeq é possivel realizar o serviço em diferentes coberturas.
Entregamos os dados brutos do sequenciamento (FastQ) e podemos realizar análises de bioinformática específicas de acordo com necessidade do cliente.
Bioinformática
O time de bioinformática é responsável por coletar, processar, analisar e interpretar dados biológicos, especialmente aqueles relacionados a sequências de DNA.
Análise de Dados Genômicos: Interpretam sequências de DNA, identificando genes, variantes genéticas e características específicas dos organismos, como patógenos em alimentos ou características genéticas.
Exemplos: 1) Sequenciamento de amplicon – identificação taxonômica; 2) Genoma Completo – montagem e anotação dos genes; 3) Sorotipagem; 4) Clonalidade; 5) Alfa e Beta diversidade.


Dúvidas frequentes:
Amplicon:
Objetivo: Identificar todos os organismos presentes na amostra, usando marcadores genéticos.
Tipo de alvo: Regiões altamente diversas do DNA (ex. 16S) que identificam gêneros ou espécies.
Vantagens: Maior número de alvos, revelando diferenças entre grupos de amostras.
Técnica: Amplificação de marcador e sequenciamento.
Tipo de resultado: Abundância relativa de micro-organismos.
Enriquecimento: Depende do ensaio.
Genoma Completo:
Objetivo: Identificar espécies/isolados ao nível de genoma completo.
Tipo de alvo: Todo o genoma do micro-organismo.
Vantagens: Metodologia padrão para identificação e caracterização de isolados.
Técnica: Fragmentação, sequenciamento e montagem do genoma.
Tipo de resultado: Caracterização da espécie, resistência a antibióticos, etc.
Enriquecimento: Não necessário.
PCR:
Objetivo: Detectar um organismo específico (ex. Salmonella).
Tipo de alvo: Gene específico da espécie.
Vantagens: Máxima precisão em amostras complexas.
Técnica: PCR em Tempo Real de marcadores específicos.
Tipo de resultado: Presença/ausência do micro-organismo.
Enriquecimento: Necessário em alguns casos (Listeria, Salmonella).
O sequenciamento de DNA em larga escala realizado pela Neoprospecta permite identificar a diversidade de bactérias e/ou fungos em uma amostra por meio da análise do gene 16S rRNA e/ou ITS rRNA. Essa metodologia oferece uma visão ampla dos micro-organismos presentes. Com um pipeline de bioinformática que melhora a resolução dos dados. Os resultados mostram alta consistência e reprodutibilidade, independentemente das variáveis técnicas analisadas, garantindo perfis microbiológicos confiáveis.
Em contratações pontuais (spot) os dados do perfil microbiológico, ferramenta dentro da Plataforma Neobiome, ficam disponíveis por 6 meses. Os gráficos gerados pela ferramenta podem ser amplamente utilizados para avaliação do resultado e geração de conhecimento e o BIM - banco de informações de microrganismos pode ser contratado para avaliação das características dos microrganismos encontrados.
Os dados brutos ficam disponíveis por aproximadamente 90 dias a partir do envio do link para download. Recomenda-se que o cliente faça o download o quanto antes, pois após esse prazo os dados expiram. Se o prazo expirar, é possível solicitar a geração de um novo link mediante uma nova proposta comercial, devido aos custos envolvidos.
A identificação ao nível de gênero ocorre quando múltiplas espécies têm similaridades nas sequências, impossibilitando a especificação.
Porém a Neoprospecta desenvolveu outros testes que utilizam primers para alvos específicos que aumentam a resolução a nível de espécie.
A grande vantagem de primers genéricos como o 16S, são amostras com microbiota desconhecida "às cegas" terem um grande direcionamento a nível de conhecimento sobre a composição microbiológica da amostra para tomadas de decisões.
Amostras com desconfiança prévia de alvos bacterianos ou fúngicos, devem ser informadas ao time comercial para seleção dos melhores ensaios para o objetivo da avaliação.
A Neoprospecta utiliza seus próprios bancos de dados validados para análises de sequenciamento, criados pela equipe de bioinformática a partir de bases públicas como GenBank e SILVA. Nessas bases, sequências com erros ou quiméricas são removidas. Para análises de bactérias (gene 16S rRNA), também estão disponíveis os bancos públicos SILVA e Greengenes, caso o cliente solicite. No entanto, os bancos públicos não passam pela curadoria da Neoprospecta, e a empresa não garante a qualidade dos resultados quando utilizados.

